Packungsgrößen (2)
| Code | Packungsgröße | Einzelpreis | Boxpreis pro Stück | |
|---|---|---|---|---|
| Produktnr. & Packungsgröße | Einzelpreis | |||

|
Code
A2156,0500
|
Packungsgröße
500 ml
|
Einzelpreis
Stück
66,20€
|
Boxpreis pro Stück
|

|
Code
A2156,1000
|
Packungsgröße
1 L
|
Einzelpreis
Stück
93,70€
|
Boxpreis pro Stück
|
Anfrage
##product_detail_publications##
-
Biochemikalien für das Labor
- Dokumenttyp
- InfoPoint
- Sprache
- de
Technische Daten
- Schmelzpunkt:
- 2,5°C
- Siedepunkt:
- 210,5°C
- Dichte:
- 1,134 kg/l (20°C)
- Brechungsindex:
- n20/D 1,4472
- Physikalische Daten:
- flüssig
- Produktnummer:
- A2156
- Produktname:
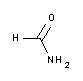
- Formamid deionisiert (Reag. USP, ACS) zur Analyse, Molekularbiologie
- Headline Kommentar:
- • Verwendung nur für den Forschungs- und Laborgebrauch!
- Spezifikation:
- DNasen/RNasen/Proteasen: nicht nachweisbar
Gehalt (GC): min. 99,5 %
Identität (IR): entspricht
Gefrierpunkt: 2,0 - 3,0°C
Farbe: max. 10 APHA
Dichte (d 20°C/4°C): 1,132 - 1,135
Ameisensäure (HCOOH): max. 0,02 %
Wasser: max. 0,1 %
Chlorid: max. 0,00005 %
Fe: max. 0,00001 %
Pb: max. 0,00001 %
Be: max. 0,00005 %
Ge: max. 0,00005 %
Mo: max. 0,00005 %
Sn: max. 0,0005 %
Al: max. 0,00005 %
Tl: max. 0,00005 %
B: max. 0,00005 %
Sr: max. 0,00005 %
Li: max. 0,00005 %
Zn: max. 0,0001 %
Mg: max. 0,0001 %
Cd: max. 0,0001 %
Pt: max. 0,00005 %
Ca: max. 0,0005 %
In: max. 0,00005 %
Sb: max. 0,00005 %
S: max. 0,00005 %
Ga: max. 0,00005 %
Au: max. 0,00005 %
Mn: max. 0,0001 %
Ni: max. 0,0001 %
Bi: max. 0,00005 %
Si: max. 0,00005 %
P: max. 0,00005 %
Cr: max. 0,0001 %
Co: max. 0,0001 %
Na: max. 0,002 %
Cu: max. 0,0001 %
Ag: max. 0,00005 %
K: max. 0,002 %
Hg: max. 0,00005 %
Ti: max. 0,00005 %
Ba: max. 0,00005 %
Zr: max. 0,00005 %
V: max. 0,00005 %
- Gefahrenpiktogramme
-
- WGK:
- 1
- Lagerung:
- RT
- Signalwort:
- Gefahr
- GHS Symbole:
- GHS08
- H-Sätze:
- H351
H360D
H373
- P-Sätze:
- P260
P280
P308+P313
P314
P405
P501
- EINECS:
- 200-842-0
- HS:
- 29241900
- Index Nr.:
- 616-052-00-8







